Bei der Durchführung genetischer Studien stoßen wir häufig auf unzureichende RNA-Proben, beispielsweise für die Untersuchung winziger anatomischer Mundtumoren, sogar Einzelzellproben, und Proben spezifischer Genmutationen, die in menschlichen Zellen auf sehr niedrigem Niveau transkribiert werden.Wenn die Abstriche für den COVID-19-Test nicht an der richtigen Stelle oder nicht genug Zeit während der Probenahme sind, wird die Stichprobengröße natürlich sehr gering sein, weshalb die Kommission für Gesundheit und Familienplanung vor zwei Tagen herausgekommen ist und hat den Test bestanden, und wenn der Nukleinsäure-Probenehmer keine sechs Proben entnommen hat, können Sie dies melden.
Die Empfindlichkeit des Reagenzes ist wichtig, weil wir dieses oder jenes Problem haben. Was können wir also tun, um die Empfindlichkeit der RT-PCR zu verbessern?
Bevor wir mögliche Lösungen diskutieren, lassen Sie uns zwei große Komplikationen mit der gerade erwähnten Situation erwähnen.
Zunächst einmal machen wir uns Gedanken über den RNA-Verlust, wenn wir nur wenige Zellpopulationen in unserer Probe haben.Wenn herkömmliche Trenn- und Reinigungsmethoden verwendet werden, wie z. B. die Säulenmethode oder die Nukleinsäure-Präzipitationsmethode, besteht eine hohe Wahrscheinlichkeit, dass die wenigen Proben verloren gehen.Eine Lösung besteht darin, ein Trägermolekül wie tRNA hinzuzufügen, aber selbst dann gibt es keine Garantie dafür, dass unser Wiederherstellungsexperiment in Ordnung ist.
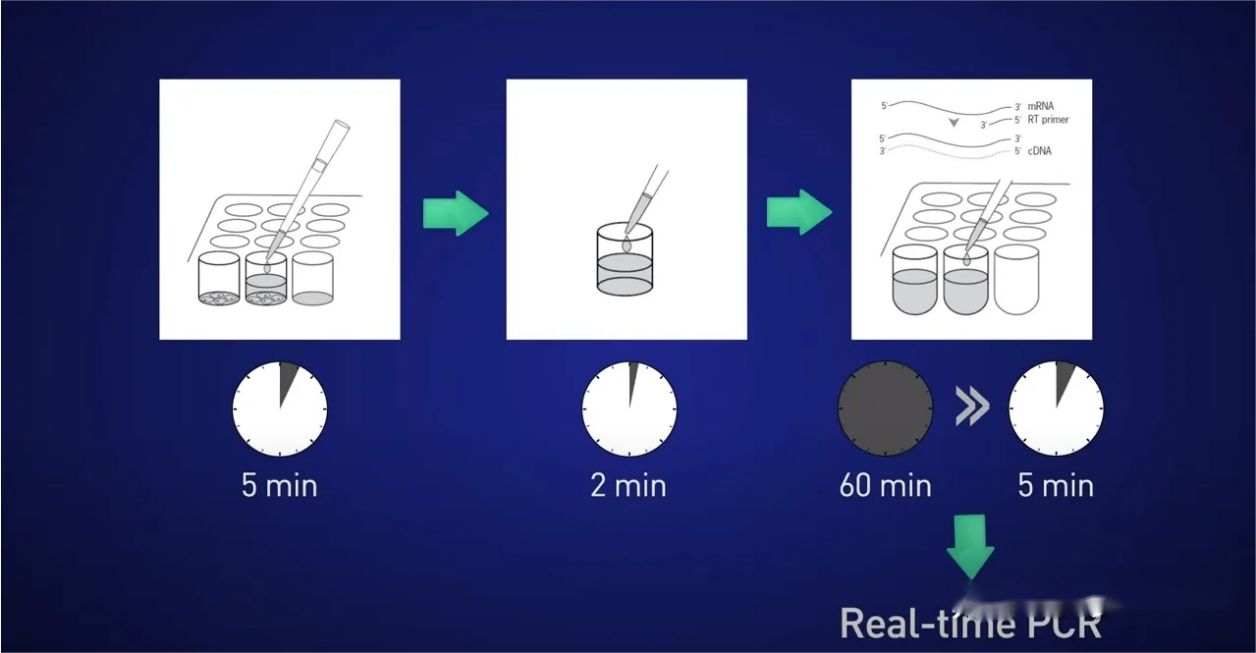
Was ist also ein besserer Weg?Eine gute Option für kultivierte Zellen oder mikroanatomische Proben ist die direkte Lyse.
Die Idee ist, die Zellen für 5 Minuten zu teilen, die RNA in die Lösung freizusetzen, dann die Reaktion für 2 Minuten zu stoppen, dann das Lysat direkt zur reversen Transkriptionsreaktion zuzugeben, damit keine RNA verloren geht, und schließlich die resultierende cDNA direkt zuzugeben in die Echtzeitreaktion.
Aber was ist, wenn wir aufgrund eines begrenzten Ausgangspunkts oder einer geringen Menge an Zielgenexpression die gesamte RNA recyceln können und immer noch nicht genügend Templates bereitstellen, um ein gutes Echtzeitsignal zu erhalten?
In diesem Fall kann der Vorverstärkungsschritt sehr nützlich sein.
Das Folgende ist ein Schema zur Erhöhung der Empfindlichkeit nach reverser Transkription.Bevor wir beginnen, müssen wir nachgeschaltet fragen, an welchen Zielen wir interessiert sind, um spezifische Primer für diese Ziele für die Voramplifikation zu entwerfen.
Dies kann erreicht werden, indem ein gemischter Primer mit bis zu 100 Primerpaaren und einem Reaktionszyklus von 10 bis 14 Mal erstellt wird.Daher wird ein speziell für diese Anforderung entwickelter Master-Mix benötigt, um die erhaltene cDNA vorzuamplifizieren.
Der Grund für die Festlegung der Anzahl der Zyklen zwischen 10 und 14 liegt darin, dass diese begrenzte Anzahl von Zyklen die Zufälligkeit zwischen den verschiedenen Zielen gewährleistet, was für Forscher, die quantitative molekulare Informationen benötigen, von entscheidender Bedeutung ist.
Nach der Voramplifikation können wir eine große Menge an cDNA erhalten, sodass die Nachweisempfindlichkeit am Back-End erheblich verbessert wird, und wir können die Probe sogar verdünnen und mehrere Echtzeit-PCR-Reaktionen durchführen, um mögliche Zufallsfehler zu eliminieren.
Postzeit: 11. April 2023